ARTICULO DEL MES DE ABRIL DE 2021
“Breast Cancer Risk Genes, Association Analysis in more than 113,000 women”
Breast Cancer Association Consortium
Con exámenes genéticos para cáncer de mama de predisposición hereditario ya de uso masivo, se han creado paneles genéticos más grandes con genes que tienen una asociación débil para riesgo de cáncer de mama. Es por eso que este estudio decide realizar el análisis del ADN de línea germinal de un panel de 34 genes para susceptibilidad para cáncer de mama de 60.00 mujeres con cáncer de mama y las comparo con 53.000 controles que participaron en estudios del Consorcio de la Asociación de Cáncer de Mama (BCAC).
Métodos
Se incluyeron 44 estudios que comparan mujeres con cáncer de mama (60.466) versus controles (53.461) del Consorcio de la Asociación de Cáncer de Mama (BCAC).
- 30 estudios basados en la población (48.826 pacientes vs 50.703 controles)
- 14 estudios basados en la familia
Se analizaron 34 genes susceptible de cáncer de mama en un panel de genes
Análisis
- Variantes de truncamiento de proteínas
- Variantes “missense” o de sentido erróneo poco frecuentes (frecuencia <0.001)
- Variantes de sentido erróneo se evaluaron según el dominio y la clasificación de patogenicidad.
- Se realizaron análisis según el subtipo de tumor, la edad y la ascendencia (europeo frente a asiático).
- Se analizaron separados para los estudios basados en la población y los estudios basados en la familia.
Resultados
- Riesgo general de cáncer de mama:
En los 5 genes (ATM, BRCA1, BRCA2, CHEK2 y PALB2) se asociaron con un riesgo significativo de cáncer de mama en general (P <0,0001) con un OR entre 2,10 y 10,57. En CHEK2, la mutación c.1100delC que correspondía al 80% de las variantes de proteínas truncas no difería el tamaño de su efecto, comparado con otras variantes de truncamiento de proteínas en este gen. Para los siguientes genes (BARD1, RAD51C, RAD51D y TP53) hubo una asociación de evidencia más modesta y la probabilidad de falso descubrimiento bayesiano fue inferior a 0,05.
- Riesgo de subtipos de tumor:
De los 12 genes con asociación a cáncer de mama, los genes ATM y CHEK2 se asocia con mayor fuerza con lo RE (+). El CHEK2 se asocian con los RE (-) y no con receptores triple negativo.
En comparación los genes BARD1, BRCA1, BRCA2, PALB2, RAD51C y RAD51D, las razones de probabilidad fueron más altas para el cáncer de mama ER (-), con una P>0,05.
• Los genes BARD1, BRCA1, BRCA2 tienen mayor asociación con ca de mama triple negativo
• En BRCA1, BRCA2 y PALB2, OR fue más altos para los tumores invasivos que para los tumores in situ.
• En contraste para ATM y CHEK2, OR para tumores invasivos y tumores insitu fueron similares
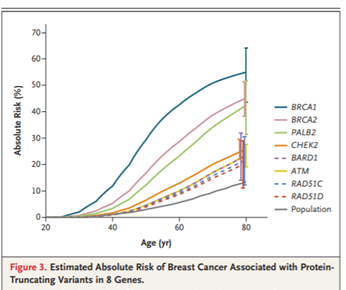
- Efecto de la edad y el riesgo absoluto:
En los estudios basados en la población el OR bajó significativamente a medida que aumentaba la edad en 6 genes BRCA1, BRCA2, CHEK2, PALB2, PTEN y TP53. Las portadoras de variantes de truncamiento BRCA1, BRCA2 y PALB2, los riesgos absolutos a los 80 años excedieron el umbral del 30% se catalogaron como de alto riesgo y a las portadoras de ATM, BARD1, CHEK2, RAD51C y RAD51D, el riesgo absoluto estimado a los 80 años se encontró en un rango del 17 a 30% catalogado como riesgo moderado según el National Institute for Health and Care Excellence Guidelines.
- Variantes “missense” poco frecuentes:
Se descubrió evidencia de una asociación con cáncer de mama en general para variantes missense poco frecuentes en 6 genes: CHEK2, ATM, TP53, BRCA1, CDH1 y RECQL.
De estos 6 genes, 2 de ellos, CHEK2 y CDH1 presentaron una asociación más fuerte con el cáncer de mama RE (+) que con el cáncer de mama RE (-) y BRCA1 demostró una asociación más fuerte con RE (-).
Discusión y conclusiones:
En este estudio involucrando a más de 60.000 pacientes y 53.000 controles permitió cálculos de riesgo estimados más precisos que lo obtenido previamente.
Se encontró una evidencia fuerte para el riesgo de cáncer de mama para variantes de truncamiento de proteínas de 9 genes con una p=0,0001 para 5 genes (ATM, BRCA1, BRCA2, CHEK2 y PALB2) y una P < 0,05 para 4 genes (BARD1, RAD51C, RAD51D y TP53).
Las variantes de truncamiento de proteínas en ATM y CHEK2 se asociaron más fuertemente con la enfermedad RE (+) y las variantes de truncamiento de proteínas en BARD1, BRCA1, BRCA2, PALB2, RAD51C y RAD51D fueron más fuertemente asociado con RE (-).
De 19 genes con un OR <2.0 (95% CI) con el umbral de 2.0 para clasificar como “alelos patogénicos de riesgo moderado”, se concluye que estos genes no son informativos para predecir riesgo para cáncer de mama. En cambio, sí se confirman una vez más que los genes BRCA1, BRCA2 y TP53 están asociadas con riesgos clínicamente significativos y las variantes CHEK2 y ATM confieren un riesgo moderado.
A considerar también está que los estudios basados en población frente a asociación familiar son mejores, ya que no sesgan la incidencia y no sobreestiman el riesgo. Además, análisis realizados en clínicas genéticas pueden tener sesgos, ya que portadores previamente identificados podrían ser excluidos de ser nuevamente testeados, por lo que es más confiable los estudios poblacionales.
Como conclusión general, las estimaciones de riesgo absoluto colocan las variantes de truncamiento de proteínas en BRCA1, BRCA2 y PALB2 en la categoría de alto riesgo y colocan las variantes de truncamiento de proteínas en ATM, BARD1, CHEK2, RAD51C y RAD51D en la categoría de riesgo moderado y nunca se debe olvidar que se deberá sumar los factores de riesgo y estilo de vida que modificaran las estimaciones.
A pesar del tamaño de este estudio, evidencia de asociación para varios genes como FANCM, MSH6 y NF1 aún no está claro. Incluso genes con claras asociaciones de riesgo a cáncer de mama, los intervalos de confianza en este estudio son amplios. Para mejorar la precisión de las estimaciones de riesgo, información del pedigrí familiar además de análisis combinados de otros estudios puede aportar.
Por este motivo, es clave la asesoría genética y los riesgos que conocemos de los genes por sí sólo, son sólo una parte de la evaluación de riesgo, un pedazo del puzle de lo que significa el riesgo de cáncer de mama. Por mientras esta información en conjunto con la evaluación de riesgo realizado en la asesoría genética, puede ayudarnos a navegar los casos clínicos y definir manejo médico de pacientes y familias de alto riesgos que enfrentamos a diario en el mundo de paneles de secuenciación masiva.
EU Maria José Mullins – Asesora genética, Centro de la mama. Clínica Alemana de Santiago.
Dr. Ítalo Tapia Tenorio, Residente cirugía UDD - Clínica Alemana
Dr. Fernando Cadiz Val, Jefe Centro de la mama. Clínica Alemana de Santiago.
